ВВЕДЕНИЕ
С декабря 2019 г. повсеместно распространяется новая коронавирусная инфекция. До сих пор ведутся активные разработки терапевтических средств для ее лечения. Известно, что заболеваемость и тяжесть течения COVID-19 варьирует в различных группах населения. Для выявления факторов риска проводились многочисленные исследования, в ходе которых были отмечены ассоциации тяжести течения новой коронавирусной инфекции с такими характеристиками, как пол, возраст, сопутствующие заболевания и генетические факторы. Важную роль в этом плане играют также гены, напрямую или опосредованно определяющие реакции иммунного ответа. Например, было показано, что варианты генов, связанных с механизмом проникновения SARS-CoV-2, и генов, связанных с врожденным иммунным ответом хозяина, являются важнейшими детерминантами тяжести течения коронавирусной инфекции [1–3].
Рецепторы врожденного иммунитета, относящиеся к NOD-подобным (NOD-like receptor, NLR) и RIG-I-подобным рецепторам (RIG-I-like receptor, RLR), имеют большое значение в развитии COVID-19. Они входят в группу паттерн-распознающих рецепторов, чья основная функция – узнавание чужеродных молекулярных структур, а также запуск сигнальных путей, приводящих к экспрессии цитокиновых и интерфероновых генов, что обусловливает последующие развитие воспаления и активацию реакций врожденного иммунитета. В частности, рецепторы типа MDA5 из группы RLR способны распознавать двуцепочечную РНК вируса SARS-CoV-2. Основными лигандами рецепторов из группы NLR являются бактериальные паттерны. Роль этих рецепторов при COVID-19 обусловлена тем, тяжелое течение заболевания и возникновение серьезных осложнений зачастую бывает связано со вторичным присоединением бактериальной инфекции. Кроме того, известно, что рецептор TLR2 способен распознавать белок S вируса SARS-CoV-2 и активировать путь NF-κB индукции воспалительных цитокинов и хемокинов. Этот путь может способствовать развитию «цитокинового шторма» и тяжелому течению заболевания [4]. Было обнаружено, что каскад реакций от рецепторов NOD2 ингибирует сигнальные пути от TLR2 и, следовательно, приводит к снижению экспрессии провоспалительных цитокинов [5].
На данный момент генетические аспекты рецепторов врожденного иммунитета в связи с коронавирусной инфекцией практически не исследованы. Изучение ключевых полиморфных маркеров в генах NLR и RLR может поспособствовать более глубокому пониманию патогенеза заболевания и усовершенствованию прогностических алгоритмов и методов его лечения. Поэтому целью настоящего исследования стало изучение ассоциации полиморфных маркеров в генах рецепторов врожденного иммунитета NOD1, NOD2 и MDA5 с развитием и тяжестью течения новой коронавирусной инфекции.
МАТЕРИАЛ И МЕТОДЫ
В исследование было включено 177 человек в возрасте 18–60 лет, проживающих в Московском регионе Российской Федерации. Материал предоставлялся ГКБ им. Д.Д. Плетнева.
В группу с патологией вошли пациенты, находящиеся в стационаре с подтвержденным диагнозом «коронавирусная инфекция, вызванная вирусом COVID-19» (вирус подтвержден лабораторным тестированием) с тяжелым и средним течением заболевания. Группу сравнения составили пациенты с бессимптомным и легким течением заболевания, а также лица из контрольной группы (здоровые люди, контактировавшие с больными коронавирусной инфекцией, но при этом ни клинически, ни лабораторно не заболевшие COVID-19).
Критериями исключения из исследования были выраженные врожденные дефекты, хронические сопутствующие патологии легких, почек, сердечно-сосудистой и нервной системы, психиатрические заболевания, метаболические нарушения, злокачественные образования, ВИЧ-инфекция, гепатиты В и С, иммуносупрессивные и иммунодефицитные заболевания, хроническое злоупотребление алкоголем и/или употребление наркотиков, беременность и/или лактация, прием препаратов иммуноглобулина или прохождение процедуры переливания крови в течение последних 3 мес до начала исследования, неспособность соблюдения пациентом условий протокола. Для включения в исследование все исследуемые подписывали информированное согласие.
У всех пациентов был произведен забор венозной крови в вакуумные пробирки с ЭДТА. После поступления в лабораторию из полученных образцов была выделена ДНК (набор «РИБО-сорб» производства «ИнтерЛабСервис», Россия). Качество нуклеиновой кислоты проверяли на спектрофотометре видимого диапазона NanoDrop (Thermo Fisher Scientific, США). Выделенную ДНК хранили при температуре -70˚С. Затем выполняли анализ ДНК с использованием метода полимеразной цепной реакции в режиме реального времени (ПЦР-РВ) на полиморфные маркеры rs2075822 в гене NOD1, rs3135499 и rs8057341 в гене NOD2, а также rs1990760 в гене IFIH1 (специально синтезированные праймеры и зонды, «Набор реагентов для проведения ПЦР-РВ», «Синтол», Россия).
В рамках статистической обработки результатов анализ частоты встречаемости аллелей и генотипов в исследуемых группах рассчитывали при помощи критерия χ2. Для количественной оценки связи между возникновением заболевания и носительством неблагоприятного полиморфного маркера рассчитывались отношение шансов (ОШ) и 95% доверительный интервал (ДИ).
РЕЗУЛЬТАТЫ
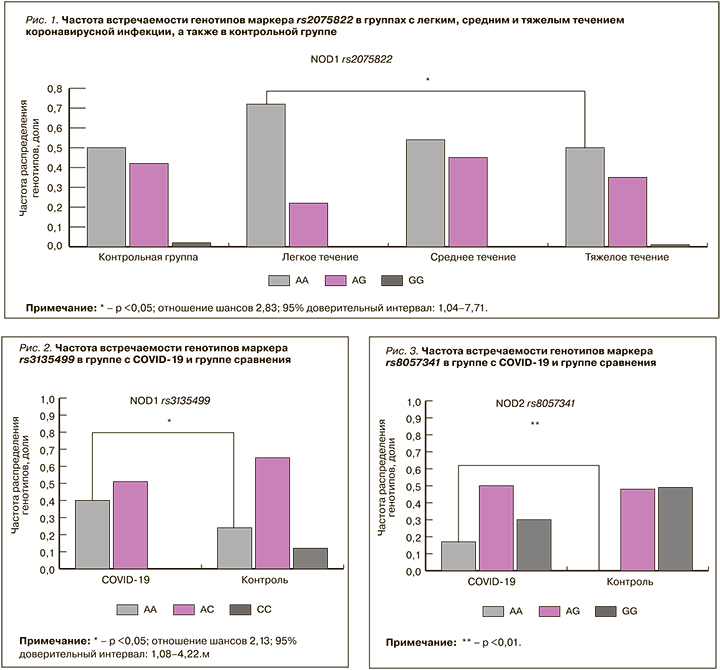
В результате проведенного исследования было выявлено, что полиморфный маркер rs2075822 в гене NOD1 не имеет каких-либо ассоциаций с риском заболеть COVID-19. Как у пациентов с COVID- 19, так и у здоровых лиц аллель A полиморфного маркера rs2075822 встречался значительно чаще: частота его встречаемости составила 0,709 и 0,802 соответственно. Исследование генотипов также не показало никаких ассоциативных связей с заболеваемостью COVID- 19. В то же время были обнаружены статистически значимые различия между группами с легким и тяжелым течением COVID-19 как для частоты встречаемости аллелей (0,875 и 0,698 для аллеля A соответственно; p <0,05; ОШ 3,03; 95% ДИ: 1,22–7,5), так и для генотипа AA (0,75 против 0,515; p <0,05; ОШ 2,83; 95% ДИ: 1,04–7,71). Соответствующие данные представлены на рисунке 1.
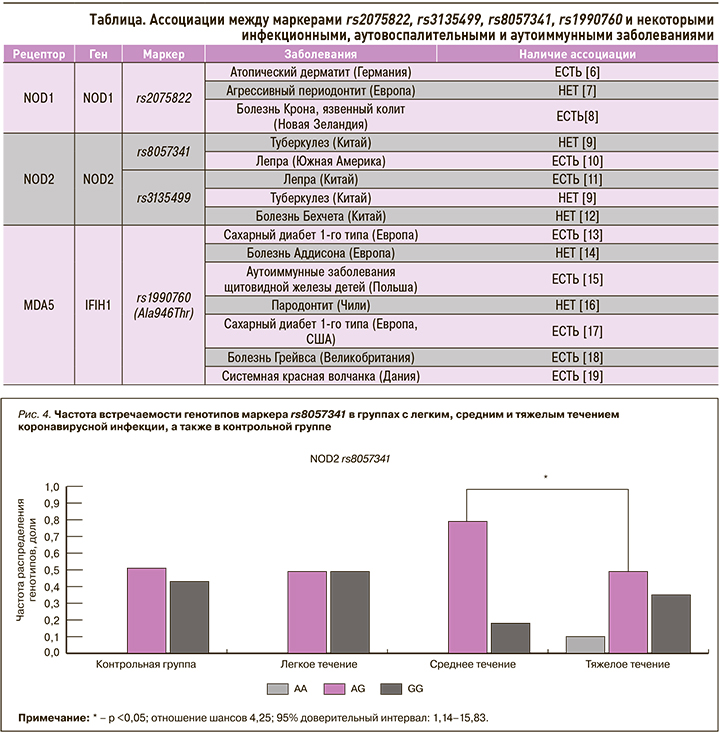
При изучении связи полиморфного маркера rs3135499 в гене NOD2 с заболеваемостью COVID- 19 было обнаружено статистически значимое различие в частоте распределения аллелей между группой с COVID-19 (частота аллеля A – 0,724, аллеля C – 0,276) и группой сравнения (0,68 – аллеля A и 0,32 – аллеля C; p <0,05; ОШ 1,6; 95% ДИ: 1–2,57). Также было установлено отличие для генотипа AA: в выборке пациентов с коронавирусной инфекцией его частота составила 0,448, тогда как в группе сравнения лишь 0,276 (p <0,05; ОШ 2,13; 95% ДИ: 1,08–4,22; рис. 2). Достоверных различий в частоте распределения генотипов AC и CC в группах не наблюдалось. Также не было обнаружено никаких ассоциаций исследуемых генотипов с тяжестью течения заболевания.
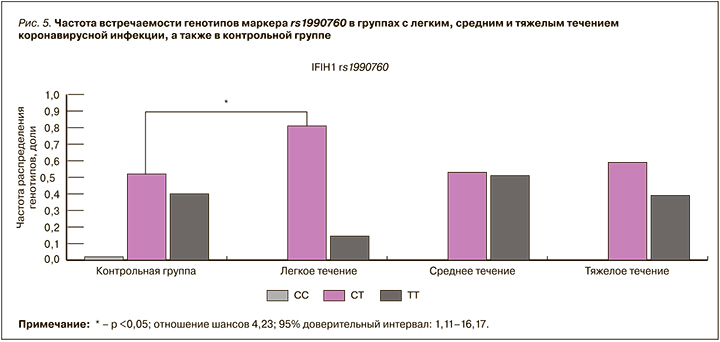
У пациентов из групп патологии и сравнения в ходе исследования маркера rs8057341 в гене NOD2 были выявлены статистически достоверные различия для аллелей. Для этих выборок частоты распределения аллелей A и G составили соответственно 0,378 и 0,621 для коронавирусной инфекции, 0,254 и 0,746 – для группы сравнения. При анализе генотипов было установлено, что гомозигота AA встречалась у пациентов с COVID-19 с частотой 0,104, тогда как в группе контроля этот генотип обнаружен не был (p <0,01; рис. 3).
Исследование также показало преобладание генотипа AG как в группе с COVID-19 средней тяжести (частота 0,813), так и в группе пациентов с тяжелым течением заболевания (частота 0,505; рис. 4). При этом различия между частотами оказались статистически значимыми (p <0,05; ОШ 4,25; 95% ДИ: 1,14–15,83).
Также для гена NOD2 мы проанализировали частоту встречаемости гаплотипов и обнаружили, что гаплотип AAAA был ассоциирован с заболеваемостью: он достоверно чаще встречался в группе больных COVID- 19 относительно группы сравнения (частота 0,1 и 0,0 соответственно; p <0,05). Для других гаплотипов достоверных ассоциаций выявлено не было.
Наконец, нами был проведен анализ ассоциаций полиморфного маркера rs1990760 в гене IFIH1 с заболеваемостью и тяжестью течения инфекции. Было показано преобладание аллеля T как в группе сравнения (частота 0,623), так и у пациентов, пораженных COVID-19 (0,705). Таким образом, ни аллели, ни генотипы полиморфного маркера rs352140 не были ассоциированы с развитием новой коронавирусной инфекции. Вместе с тем были выявлены статистически значимые отличия в распределении генотипов исследуемого маркера у группы пациентов с легким течением заболевания (частота встречаемости генотипа CT 0,833) по сравнению с контрольной группой (частота 0,542; p <0,05; ОШ 4,23; 95% ДИ: 1,11–16,17; рис. 5).
ОБСУЖДЕНИЕ
Однонуклеотидные полиморфизмы (Single Nucleotide Polymorphism, SNP) представляют собой отличия последовательности ДНК размером в один нуклеотид в геноме представителей одного вида или между гомологичными участками гомологичных хромосом. SNP могут влиять на восприимчивость к заболеванию, его патогенез и эффективность терапии.
SNP в генах рецепторов врожденного иммунитета приводят к изменению уровней экспрессии провоспалительных цитокинов и хемокинов при различных патологиях. Нами был проведен анализ литературных данных, в ходе которого было обнаружено, что маркеры rs2075822, rs3135499, rs8057341 и rs1990760 ассоциированы с некоторыми инфекционными, аутовоспалительными и аутоиммунными заболеваниями (табл.), однако в контексте коронавирусной инфекции эти маркеры изучены еще не были.
В ходе представленного исследования мы осуществили сравнительный анализ распределения аллелей, генотипов и гаплотипов в полиморфных маркерах rs2075822 в гене NOD1, rs3135499 и rs8057341 в гене NOD2, а также rs1990760 в гене IFIH1 в группе с коронавирусной инфекцией и группе сравнения. Было продемонстрировано, что выбранные нами маркеры ассоциированы с заболеваемостью коронавирусной инфекцией: наличие аллеля А rs3135499 в гене NOD2 повышает риск заболеть в 1,6 раз, генотипа АА – в 2,13 раз. В случае маркера rs8057341 аллель А повышает риск заболеваемости в 1,78 раз. Также гаплотип AAAA был ассоциирован с риском возникновения новой коронавирусной инфекции.
Кроме того, сравнительный анализ был проведен в группах пациентов с разной степенью тяжести течения COVID-19 и в контрольной выборке. Было установлено, что наличие генотипа CT маркера rs1990760 в гене IFIH1 повышает вероятность легкого течения заболевания в 4,23 раза. Легкое течение также было ассоциировано с маркером rs2075822 гена NOD1: аллель A увеличивает его вероятность в 3,03, а генотип AA – в 2,83 раза. Наконец, генотип AA rs8057341 в гене NOD2 повышает вероятность протекания COVID-19 средней тяжести в 2,83 раза.
ЗАКЛЮЧЕНИЕ
Таким образом, данные нашего исследования могут послужить в качестве предиктивных маркеров при прогнозировании тяжести течения коронавирусной инфекции у здоровых лиц с неблагоприятным анамнезом и склонностью к легочным заболеваниям, а также для раннего выявления больных, предрасположенных к тяжелому течению COVID-19, среди всех пациентов с уже развившимися клиническими проявлениями инфекции. Полученные результаты могут поспособствовать принятию превентивных мер еще до наступления тяжелых клинических проявлений COVID-19. Кроме того, возможно их использование в дальнейших исследованиях в области персонифицированной медицины.



